動物中DNA轉座子通過兩種機制介導基因重復
轉座子被認為是宿主基因組演化的重要推動力。其類型眾多,包含non-LTR(Long Terminal Repeat)型逆轉座子、LTR型逆轉座子、Helitron型DNA轉座子、TIR(Terminal Inverted Repeat)型DNA轉座子等,可引起包含基因重復(gene duplication)在內的各種遺傳突變。已有研究表明,non-LTR型逆轉座子在人類基因組中介導基因重復,產生大量新基因。2016年,中國科學院院動物研究所研究員張勇研究組研究發現,動物中LTR可借由模板跳轉介導基因重復;Helitron在蝙蝠中通過通讀(transduction)的形式介導基因重復。迄今,TIR型轉座子介導基因重復的能力只在植物中被揭示,其中水稻中的MULE元件捕獲大量宿主基因,形成Pack-MULE的嵌合結構。但TIR型轉座子介導基因重復形成的機制仍不清楚,動物中此類基因重復也鮮有報道。
7月13日,張勇研究組等在Nature Communications上,在線發表了題為DNA transposons mediate duplications via transposition-independent and -dependent mechanisms in metazoans的研究論文。該研究首次大規模搜索了100個動物基因組,鑒定了TIR轉座子介導的基因重復,并將其命名為Pack-TIR。研究發現,大部分基因重復的產生不依賴轉座活性,符合缺口填補(gap-filling)模型;在黑腹果蠅群體基因組數據中找到的最年輕的多拷貝基因Ssk-FB4則符合新提出的模板跳轉及轉座(FoSTeST)模型。蛋白質組、自然選擇壓力分析及表型關聯分析顯示Ssk-FB4s編碼功能蛋白。上述結果證明TIR轉座子可以重塑基因結構,產生新基因。
研究中,科研人員收集了公共數據庫UCSC中100個后生動物的基因組,計算分析鑒定出370個Pack-TIR元件(圖1A)。進而,同時在黑腹果蠅群體中鑒定出一個多拷貝Pack-TIR家族:Ssk-FB4(由FB4 轉座子捕獲基因Ssk所形成的新基因)。其基因結構和植物中Pack-MULE相似,中間為捕獲的外源序列,兩側為TIR轉座子序列(圖1B)。
研究發現,Pack-TIR與TIR轉座子數目成線性正相關(圖2A)。對靈長類譜系中Pack-TIR的起源時間分析發現,其零散分布在各演化枝上(圖2B)。這些Pack-TIR在各個時間段皆有起源,即使宿主編碼的轉座子已喪失轉座活性。由此推斷,大部分Pack-TIR的產生不依賴于轉座活性(transposition-independent)。多方面證據表明,Pack-TIR的形成符合缺口填補(gap-filling)模型(圖2C):Pack-TIR的豐度與其到母源序列的距離呈負相關,即TIR轉座子傾向于捕獲臨近序列;67.6%事件含有微同源序列(Pack-TIR與母源序列共享較短的相似序列);靈長類物種間的比較分析顯示,轉座子和外源序列是先后插入的兩次獨立事件。隨后,研究人員發現果蠅中Ssk-FB4其形成過程與缺口填補模型不符,因此提出復制叉停滯-模板跳轉-轉座(FoSTeST)這一新模型來解釋多拷貝Pack-TIR的產生(圖2D)。該模型與缺口填補模型不同,模板跳轉后轉座酶會立即將嵌合的Pack-TIR轉座到基因組其它位置,科研人員的策略及發現對植物中Pack-MULE的起源機制研究將具有啟發意義。
除機制方面的分析,科研人員還結合轉錄組數據和進化基因組數據探索Pack-TIRs的功能。研究發現,Pack-TIR傾向于捕獲外顯子序列,且大部分與所在基因發生融合,具有轉錄信號(圖3A)。Ssk-FB4是罕見的編碼功能蛋白的例子,所有拷貝不僅顯示出較高的轉錄和翻譯水平(圖3B-C),且各拷貝的非同義突變在細胞膜外結構域顯著富集,暗示其受到正選擇(圖3D)。此外,突變機制所賦予的Ssk-FB4嵌合結構使其更容易發揮新功能,可能增加果蠅抵御環境中致病微生物的能力(圖3E-F);轉座帶來的拷貝數增加使自然選擇有更多的作用對象,推動了適應性進化更快發生。
該研究由中國科學院動物研究所、法國居里研究所、北京大學腫瘤醫院等機構合作完成。研究工作得到國家重點研發計劃、國家自然科學基金、中科院青年創新促進會等資助。
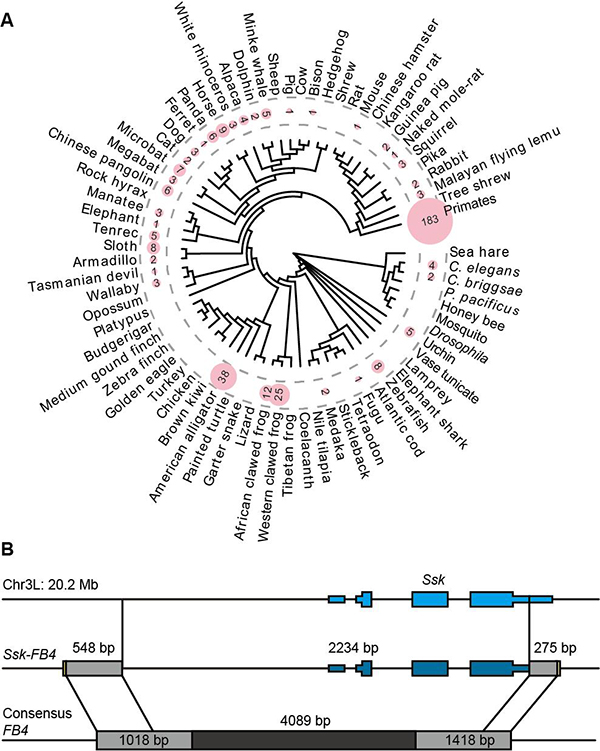
圖1.動物中Pack-TIR分布及結構示意圖。(A)370個Pack-TIR在100個動物中分布情況。圓圈內數字表示對應物種的Pack-TIR數目。(B)黑腹果蠅群體中Ssk-FB4的基因結構。藍色片段為Ssk的外顯子,黑色和灰片段為FB4序列。
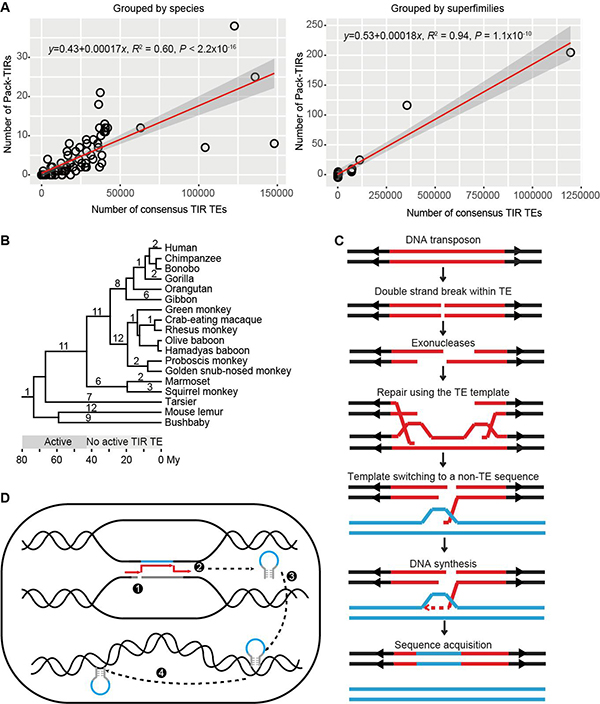
圖2.動物中Pack-TIR形成的兩種機制。(A)各物種或各轉座子超家族中Pack-TIR與TIR轉座子數目的線性關系。(B)Pack-TIR在靈長類動物演化樹上的分布,數字表示Pack-TIR出現在某一特定演化枝的個數,下方顯示了轉座子活躍與否的時間,單位為百萬年。(C)缺口填補模型示意圖。紅色片段及兩側箭頭表示為DNA轉座子,藍色片段為外源序列。該圖顯示轉座子內部雙鏈斷裂,也可能是轉座子的自主切割引發缺口。(D)FoSTeST模型示意圖。1.復制叉停滯在轉座子處并發生雙鏈斷裂;2.轉座子和母源序列空間接近,導致修復時模板跳轉產生嵌合片段;3.轉座酶識別嵌合片段,切割插入到其它位置;4.由于Ssk-FB4保留轉座子的末端反向重復序列(TIR),在轉座酶的幫助下增加其拷貝數。
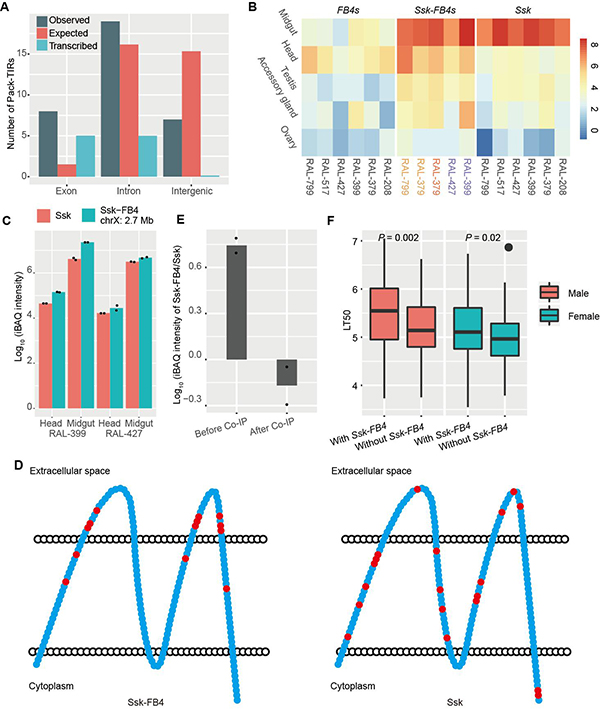
圖3.Pack-TIRs的表達、選擇和關聯分析。(A)人類中Pack-TIR捕獲序列的類型分布情況。(B)Ssk、Ssk-FB4、FB4在六個黑腹果蠅品系五個組織內的表達譜,表達值為log2(TPM值)。(C)Ssk和Ssk-FB4蛋白水平的表達量。(D)Ssk-FB4/Ssk蛋白非同義突變積累分布圖。(E)Ssk和Ssk-FB4蛋白與Mesh蛋白免疫共沉淀前后的蛋白定量數據。與母本基因Ssk不同,Ssk-FB4與Mesh互作水平較弱,暗示其具有新功能。(F)黑腹果蠅DGRP品系在真菌Metarhizium anisopliae Ma549感染后的壽命半衰期值。
-
科技前沿

-
科技前沿

-
項目成果