納米孔測序技術
測序長度和準確率的快速提升使得納米孔測序有望顛覆DNA測序市場。
紐約威爾康奈爾醫學院的計算生物學家Christopher Mason喜歡在會議上表演一個“絕活”:他和同事先從志愿者手機上收集DNA樣本,然后就能在一個小時內現場進行譜系分析,甚至敘述志愿者一天的生活細節。“我們能從留在手機上的DNA信息推測誰剛剛吃了橘子或者誰吃了豬肉。”Mason說。
Scott Tighe (左) 和 DavidGoerlitz 正在使用MinION設備,在南極的泰勒谷對微生物DNA進行測序。
圖片來源:Sarah Johnson
Mason之所以能夠進行如此快速的分析,主要得益于英國牛津納米孔技術公司(ONT)研發而成的一種手持測序設備——MinION。MinION通過將DNA長鏈穿過納米孔,探測由DNA中四種核苷酸引發的微小電流變化,從而讀取序列信息。
Mason以一種輕松隨性的方式大致展示了該設備的性能,而早期用戶已經利用它取得了一些引人矚目的科學成就。MinION在2015年監控埃博拉病毒爆發中發揮了重要作用,它曾到達過南極,甚至進入了太空。
然而大小相當于一幅撲克牌的MinION在全球測序市場上僅占據了一小部分份額。目前測序市場仍由位于加州圣迭戈的Illumina公司主導。Illumina的起步早了十年,而ONT及其用戶也在努力克服技術挑戰,特別是較高的出錯率。與此同時,競爭企業希望對這種概念上很簡單但技術上很復雜的測序策略加以創新,最終超越ONT。
艱難的開始
Illumina通用的DNA測序技術通過在DNA復制反應期間順序讀取摻入樣品中的堿基,產生大量非常準確的短讀段(reads)。然后,這些長度為幾百個核苷酸的序列數據可以通過計算組裝成含有數百萬個核苷酸的“重疊群”(contigs)。
至于納米孔測序技術,則是讓完整的DNA片段穿過納米孔,直接進行分析。
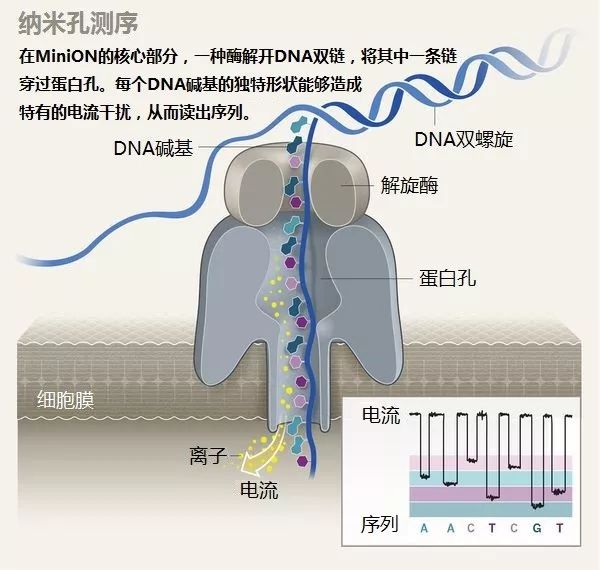
MinION的每個流通池成本為500-900美元,包含數百個納米孔,因此可以同時分析許多分子。系統在每個孔上施加電壓,隨著酶穩定地牽引DNA通過,核苷酸阻斷離子流動并產生微小的電流變化,最后由專業軟件破譯這些變化(參見“納米孔測序”一圖)。由此產生的長讀段可達數千個核苷酸,簡化了組裝過程。
Image: Nik Spencer/Nature
自2014年發布以來,MinION的潛力令基因組學界興奮不已,但早期用戶遇到了許多挑戰。“僅僅對單個細菌基因組進行測序就需要很多工作,因為產量很低,單讀段的準確性也很低。”英國伯明翰大學微生物基因組學家Nicholas Loman說, 他是MinION最早的一批使用者之一。
Illumina的單個讀段的平均準確度通常可達99.9%,而第一代MinION測定每十個堿基中就約有三個是錯的。此外還有一些其他問題。“有時候一個流通池是很好的,但另一個卻不知道因為什么原因只有三個孔能用,盡管它們都來自同一批次。”Mason說。ONT不愿意對這篇文章公開發表評論。
這些限制使得MinION的應用主要集中在以快速簡單為至高要求的應用上,如病原檢測。美國國家人類基因組研究所(US National Human genome Research Institute)基因組信息學負責人 Adam Phillippy表示:“如果想知道一封信件中是否含有炭疽桿菌,用MinION很快就能做到,盡管讀段不完全準確。”
飛速的發展
Keith Robison是馬薩諸塞州藥物發現公司Warp Drive Bio的首席科學家,他表示現在的MinION已經成熟。更重要的是,ONT頂住了產品發布后受到的懷疑和不屑。Robison說:“MinION已經多次證明所有這些人都錯了。”
源自大腸桿菌的孔蛋白以及流通池化學的進步已經將許多實驗中單個讀段的錯誤率降低至2-5%。數據輸出的大幅提升使得研究人員能夠通過同時對更多分子進行測序,更好地發現錯誤,并且讀段長度從最初的約7,000個核苷酸躍升至今天的100,000個核苷酸。
Loman的團隊已經將單讀段長度增加到了近一百萬個堿基。“在早期運行中,我們每次運行都能測定幾百Mb。”加拿大安大略省癌癥研究所的生物信息學家Jared Simpson說,“通過新的納米孔,測序長度很快達到了Gb級別,現在每次運行可達5 Gb或10 Gb。”
該平臺還受益于軟件開發的迅猛發展。由于存在更多重疊,將長讀段組裝為重疊群比短讀段容易,但納米孔讀段可能出現更多錯誤,而且處理長讀段的計算量極大。
為了解決這個問題,Phillippy的小組設計了一種名為Canu的算法,在該算法中,傳統的短讀段組裝過程被作為長度短段數據的補充。另一種軟件工具——ONT的Scrappie則解決了因均聚物(包含多個連續相同核苷酸的序列,例如AAAAA)導至系統停滯而產生的測序錯誤。
適用于微生物測序
事實證明,MinION在傳染病研究人員中尤其受歡迎。Loman與同全球病毒學熱點研究區域的同事合作,共同監控埃博拉在西非以及寨卡在巴西的傳播。“他們基本上能在48小時內建立一個測序實驗室并使其運轉,裝備可以放在行李箱里攜帶上飛機。”加州大學圣克魯茲分校的生物物理學家Mark Akeson說,他開展了納米孔測序法方面的一些基礎性研究,并且是ONT咨詢委員會成員。
Loman認為這種可攜帶性是一種巨大的優勢,但他強調大量的數據輸出可能令人難以招架。“我們在巴西幾乎要成功了,但設備過熱卻導至我的Mac崩潰。”
一些團隊正在探索臨床微生物學應用。澳大利亞昆士蘭大學的生物信息學家Lachlan Coin開發了實時數據分析算法,用于檢測血液樣本中的耐藥細菌。在利用培養細菌和舊流通池進行的早期測試中,Coin團隊能在10個小時內鑒定出一個樣本中的所有抗藥基因。
Coin表示,現在的技術能讓這一時間減半,但真實樣本中人類DNA會覆蓋細菌DNA,從而使一過程復雜化。他還認為,再過一年左右,將能夠在6個小時內鑒定病人樣本中的抗藥基因。
另外一些研究人員則在探索宏基因組學,目標是全面描述樣本中的所有生物。原則上,流通池中的各納米孔可以用來同時檢測不同的基因組。“你可以獲得其中任何物種的完整基因圖譜,包括細菌、病毒和人類DNA。”Mason說。
他利用納米孔測序對臟亂的紐約地鐵系統開展了宏基因組研究,此外還有對更加惡劣的環境(包括火星)進行分析的宏偉計劃。Mason同美國宇航局的科學家合作證實,MinION在國際空間站零重力條件下表現良好。他和同事們希望有一天能將該技術帶到火星,為尋找地外生命提供幫助。
而在地球上,佛蒙特大學的遺傳學家Scott Tighe在南極麥克默多干河谷(McMurdo Dry Valleys)也運行了MinION,他的團隊用了兩個多小時對微生物樣本進行測序。“設備停止運行的原因在于外面太冷了,電池無法繼續工作。”與Tighe合作過若干項目的Mason解釋到。
遠大的目標?
Adam Phillippy等納米孔方面的專家將微生物基因組組裝視為“一個已經解決的問題”。如今,他們有了更高遠的目標:含有數十億個而非幾百萬個核苷酸的哺乳動物基因組。
2017年,包括Phillippy、Loman和Simpson在內的多機構研究團隊稱,他們僅利用MinION數據就完成了人類基因組組裝,并實現了高連續性和精確度。Simpson表示,平均的重疊群大小達到百萬堿基級別,精度值最高為99.44%。結合Illumina的短讀段技術,該團隊將準確度提升至99.96%,不過仍未達到組裝項目通常所追求的最高標準99.99%。
不過,在人類基因組分析的其他方面,納米孔技術仍有很大優勢。例如,目前的人類基因組組裝仍不完整,因為高度重復的區域無法進行短讀段分析。
Karen Miga是加州大學圣克魯茲分校的一名基因組學研究人員,她的團隊表明,納米孔能幫助研究人員填補這些空白,他們利用150千堿基對的讀段重構了人類著絲粒。此前對該領域的研究是一片空白。據與Miga合作的Akeson預測,可能再過幾年就能組裝出真正完整的基因組序列。
納米孔分析還非常適合發現表觀遺傳標記——單個核苷酸的微小化學修飾,可影響基因表達。大多數測序平臺采用的是除去這些標記的樣品制備方法,但納米孔平臺可直接分析修飾的DNA。
Simpson和來自約翰·霍普金斯大學的Winston Timp表明,他們能通過軟件區分甲基化胞苷酸和正常胞嘧啶的電信號,準確度約為90%。Akeson也成功完成了相似的工作。“我們能檢測到我們想發現的任何修飾。”Akeson表示,“它甚至能夠發現小至兩個氫原子的微小區別。”
展望未來
盡管在納米孔市場中僅此一家,但ONT仍需面對長讀段競爭者。加利福尼亞的太平洋生物科學公司(PacBio)憑借以極高的準確率測量成千上萬個堿基的DNA片段而享有盛譽。PacBio的平臺比MinION龐大且更昂貴,它最小的系統Sequel大約是冰箱的大小,售價35萬美元,此外它也不能完全達到ONT的最大讀段長度。
但PacBio的首席科學官Jonas Korlach指出,該系統能夠準確產生平均長度10-18kb堿基的讀段,最長讀段可達100kb,同時PacBio因其高質量數據而在基因組學領域受到信賴。Phillippy說:“利用Pacbio的技術很容易達到99.99%的準確率。”
該技術仍然是他進行大規模基因組組裝的首選。對于大型項目而言,它的速度更快:最近報道的利用納米孔數據組裝人類基因組耗時為PacBio的十倍。
一些用戶發現納米孔樣本制備盒可能會得到無法預期的結果,一些DNA樣本需要大量優化。“一些人用得非常好并且獲得了良好的結果,但其他人仍在苦苦掙扎。”Robison 表示。在去年12月的一次演講中,ONT首席技術官Clive Brown宣稱:“公司正在投入大量努力,來幫助用戶針對特定樣品類型改進實驗方法,從而優化結果產出。”
同樣,該公司最大的優點之一——通過定期升級優化來提高性能——可能會給某些忠實用戶帶來困擾。例如,ONT最新的納米孔就給習慣了舊技術的用戶帶來了難題。“這種情況已發生了多次,”Loman說,“這就是走在前沿的弊端,它們不會如你所愿長期保持不變。”
這些問題為競爭者制造了機會,比如瑞士的羅氏公司。2014年,羅氏收購了總部位于加州的納米孔初創公司—— Genia Technologies。雖然羅氏對其系統保密,但Genia在2016年公開的一份文件中描述了“通過合成開展納米孔測序”的策略,也即將DNA合成酶同蛋白納米孔配對。
這種酶會讀取目標DNA,并且利用帶有化學標簽的核苷酸合成互補序列。在每個堿基被添加到不斷延長的DNA鏈過程中,它的標簽被釋放并穿過納米孔,從而產生獨特的電信號。
羅氏測序方案負責人Neil Gunn表示,雖然這項研究早于羅氏收購,但該技術的核心原則在很大程度上沒有改變。“這非常符合產品的設計,”他說,“自那時起,我們一直致力于提高準確度、讀取速度和測序長度。”Gunn指出,羅氏的平臺將直接針對體外診斷領域,目標是超越競爭對手的準確度和可重復性。
?Robison認為羅氏的平臺是潛在的強有力競爭者,早期研究顯示,對于任何給定核苷酸,準確率在78%到99%之間。他表示:“他們的設備可能值得關注,但魔鬼總是在細節中,我們需要大規模地評估真實數據。”
不過,ONT也未曾止步不前。和此前的模型相比,兩個最新的臺式系統能產生更大的數據量。在2017年3月發布的GridION可同時運行多個MinION設備。相比之下,PromethION利用的是一種完全不同的流通池,并且專門為人類基因組尺度的項目而設計。
“很顯然,他們希望該系統能在輸出量方面同Illumina公司的平臺一較高下。”Loman表示。這些設備已經提供給部分用戶試用,但還沒有相關公開數據。
雖然這些進展的結果尚不可知,但納米孔測序毋庸置疑具有極大的優勢。它有望為大眾提供低成本且可靠測序結果的潛力令研究人員興奮不已。“作為計算機科學家,我總是渴望獲得更多數據。”Phillippy表示,“所有微生物實驗室,甚至大學課堂都能自己測序,想想就覺得很好。”?