orf1ab基因為何成為新型冠狀病毒核酸檢測的靶序列?
為什么當前nCov的核酸檢測試劑盒以ORF1ab、S和N作為目標基因?為什么單用ORF1b做進化就能推測nCov來源于蝙蝠?為什么用如此短的時間就能比較分析那么多種coronaviruses基因組從而否定重組陰謀論?我睡前思考著這些問題,索性搜搜文獻看。精神食糧讓我意識到自己是個無知的人類。
我檢索了有關nCov基因組可視化的文獻,截取了Paraskevis等人文章的組裝注釋圖[1](按道理講我更希望貼我國科學家發表在nature上的,無奈尺寸和配色不適用于手機屏幕),如下:
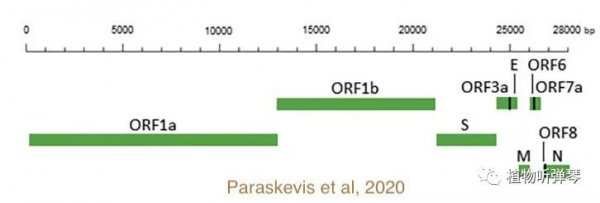
所以看看吧,這么小的基因組,就這么幾個主要的基因,你還想用哪個做qPCR?!哈哈哈,我真的是如此愚鈍。
此外,如此小的基因組,加上病毒基因組屬于單正義鏈RNA(所以也不會出現高等植物多倍體、雜合等問題),對下游計算分析的硬件要求及耗時也將相應降低,分析個重組不重組的當然會更快。
Zumla等人對冠狀病毒的生活史做了比較全面的綜述[2]。病毒借助宿主系統進行后代的基因組擴增和外殼組裝是一個最基本的戰術動作。其中ORF1a和ORF1b開放閱讀框有重疊,編碼large replicase polyprotein 1a (pp1a)和pp1ab。這倆大蛋白在被蛋白酶剁吧剁吧以后,釋放出病毒轉錄翻譯依賴的蛋白。S基因決定病毒的相貌,從英文Spike protein就可以很好地理解它的功能。N(Nucleocapsid)、M(Membrane)、E(Envelope)基因編碼病毒后期封裝和胞吐相關的結構蛋白。理論上講,選取那個基因做檢測分析,重要的還是引物的特異性要好,靈敏度要高;同時我個人理解,盡量選取變異幾率小的基因,這樣能避免試劑盒的反復開發,加快“早發現早隔離”的進度。最初Takara第一版試劑盒明確說明了對N蛋白基因特異性不太好,估計近期會有改進。Hu等人提到ORF1a/b的變異性相對較小,而S變異性略高[3]。我猜最后檢測策略應該是以ORF1a/b為主,其他基因為輔吧。
既然ORF1a/b是相對保守的,那么用ORF1b做病毒的進化分析就是相對科學的。
-
焦點事件
-
焦點事件












