三種長片段測序方法對比

近期,由澳洲昆士蘭大學發表在bioRxiv的文章《Comparison of long read methods for sequencing and assembly of a plant genome》中,采用三種長片段測序技術Pacific Biosciences (Sequel I), Oxford Nanopore (PromethION、MinION) 和華大智造stLFR(single-tube Long Fragment Read)對夏威夷果(Macadamia jansenii)進行了基因組組裝,并分別從組裝結果的準確度、完整度、測序成本及樣本要求等四個方面進行了對比。
文庫構建和上機測序
采用15ug?DNA 構建30Kb以上的文庫用于PacBio測序,并將該文庫用sequel I測序8個SMRT cell,得到317萬條subreads,總數據量為65.2Gb,N50為35.9Kb。
基于ONT平臺構建文庫的DNA投入量為1.5ug和1.2ug,將其文庫分別在PromethION和MinION測序,得到的下機數據分別為23.2Gb和1.7Gb,reads的N50分別是28.5Kb和16.6Kb。
華大智造stLFR文庫構建從10 ng DNA稀釋液中提取1.5ng用于后續建庫,然后采用MGISEQ-2000RS進行測序,得到約74.5Gb的數據用于后續組裝。
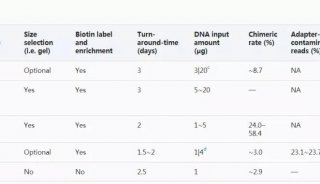
對比上述三種長片段組裝方法可知,stLFR文庫組裝所需投入量遠低于PacBio及ONT,僅需要1.5ng即可。同時,根據表1可知,基于華大智造DNBSEQ平臺的測序成本最低,不到PacBio的十分之一。
表1 測序數據統計

數據處理和分析
在對下機數據做接頭和低質量過濾后,分別得到25Gb (ONT)、65Gb (PacBio)、74Gb (MGI)數據用于后續分析。設置k-mer為21-,23-和25-對基因組大小進行評估,已知夏威夷果的基因組大小約為616-653Mb。
分別用Redbean v2.5、Flye v2.5、Canu v1.8和Raven對ONT數據進行組裝。結果顯示Canu 和Flye能得到更大的基因組和更多的contig數,其次是Redbean (~750 Mb, contig N50 ~700 kb)和Raven (~720Mb, contig N50 ~500 kb) 。Canu在32X的contigN50為1.43Mb,遠遠大于21x下的706Kb。Flye的運算速度比Canu快5倍。
用Redbean、Flye、Canu及Falcon對PacBio數據進行組裝,結果顯示Falcon結果最好,contig N50為1.38Mb,Redbean的組裝結果最分散,完整度只有89%。Flye的組裝結果更接近Falcon,但是基因組大小只有767Mb,Canu的重復序列比例太高,組裝的基因組達到1.2Gb。
使用superNova 2.0對stLFR進行組裝,得到752 Mb 的基因組,contig N50為 35.6 kb,scaffold N50 為3.54 Mb,BUSCO評估基因組完整度為88.3%,組裝結果的mismatch和Indel最少,準確性最高。
表2 基因組組裝結果

注:表中的BGI即代表采用了基于華大智造DNBSEQ平臺的stLFR技術得到的組裝結果。
進一步地,如果使用ONT數據對組裝結果進行“補洞”,則contig N50 可以從35.6Kb提升到1.05Mb,最長的contig從518 kb 提升到9.7 Mb,gap數從24,933 降低到2,284,基因組完整性也提高了4.8%,達到了92.5%。
表3 用ONT數據對stLFR組裝結果進行“補洞”

組裝結果比較
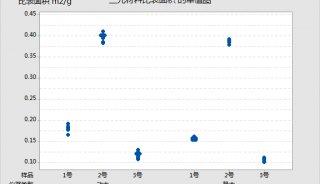
用QUAST v5.0.2對組裝結果進行評估,并與已發表的夏威夷果基因組進行比較。為了評估堿基的準確度,用QUAST計算組裝結果里的mismatch和InDel比例,結果顯示:基于華大智造stLFR技術的mismatch和InDel均是最低的,組裝結果具有最高的準確性;ONT組裝結果在校正前后有明顯的差異。

圖1 ?組裝結果堿基準確性比較
用BUSCO對各版本組裝結果進行完整性評估,采用的數據集為eudicotyledons_odb10 (2121 genes)。結果顯示:ONT+Illumina、PacBio以及stLFR+ONT三種方法的完整性無明顯差異,均達到92%以上。

圖2 組裝完成性評估
討論與結論
通過對比分析,Pacific Biosciences (Sequel I), Oxford Nanopore (PromethION) 和華大智造MGI stLFR三種長片段測序技術都能得到非常好的組裝效果。相對而言,華大智造stLFR技術所需投入量最低,僅為納克級別,并且組裝結果的mismatch和InDel最低,準確度最高,特別適用珍貴樣本或極端樣本檢測,同時測序成本上具有顯著優勢。
參考文獻:
Comparison of long read methods for sequencing and assembly of a plant genome.
doi: https://doi.org/10.1101/2020.03.16.992933