MALDI引導的空間定位組學對腫瘤亞細胞群的深度蛋白組分析

MALDI Guided SpatialOMx,即基于質譜成像定位的空間多組學分析,完美的將MALDI成像技術和傳統LC-MS/MS組學流程結合在了一起,它的特點是利用了MALDI成像技術能在分子原位對生物分子進行定位的特點,在生物組織的內部鎖定目標微區(ROI,Region of Interests),在對該目標微區實施激光微切割(LCM,laser capture microdissection)后,進行LC-MS/MS組學分析,從而實現了深度的蛋白組信息挖掘。
實驗分享
荷蘭馬斯特里赫特大學的Ron M.A. Heeren教授實驗室利用空間定位組學工作流程,對來自于乳腺癌患者的腫瘤組織,進行了蛋白質分子特征的深度研究。

Ron M.A. Heeren 教授
馬斯特里赫特多模型分子成像研究所(Maastricht MultiModal Molecular Imaging institute)質譜成像部主任,荷蘭馬斯特里赫特大學(Maastricht University)
Ron M.A. Heeren教授的實驗室側重于分子成像技術研究和與臨床相結合的應用開發,發表了超過200篇科研論文,他的H因子(H-index)高達到38。
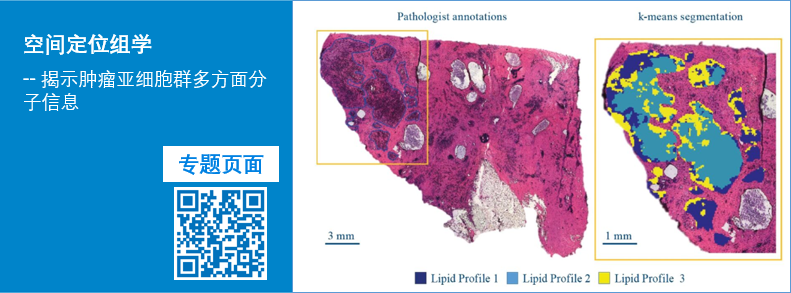
在對組織切片進行化學基質噴涂之后,利用布魯克timsTOF fleX質譜系統,針對脂質成分進行成像數據采集。在洗去基質之后,對該組織切片實施H&E染色得到如圖1所示的染色結果,用于后續的病理學評估和目標微區ROI的鎖定。
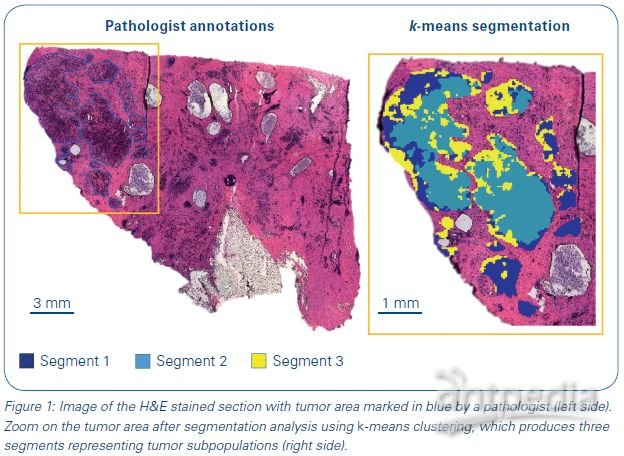
MALDI成像數據經過布魯克成像軟件SCiLS Lab 2019c的聚類分析,結合對腫瘤區域的病理學標注,確定了三個腫瘤亞細胞群的目標微區ROI,即圖1中的Segment 1-3。先由MatLab R2018a軟件對上述的微區數據進行初步的處理和優化,然后導入Leica LMD7000中,分別對三個目標微區ROI實施了激光微切割。將所獲得的細胞簇(約2000個細胞)分別進行還原、烷基化、酶切,再經過布魯克nanoElute液相系統分離,最后進入布魯克timsTOF fleX質譜儀,在PASEF模式下進行了一級質譜和二級質譜的數據采集。所獲得的數據被導入到PEAKS X中進行數據庫檢索,最后將檢索結果用基因腫瘤學軟件PANTHER V.13.1進行分析。
實驗結果表明,三個腫瘤亞細胞群表現出不同的蛋白組學特征:腫瘤亞細胞群1和2的“生物調控”和“發展進程”呈現為明顯的低表達,而腫瘤亞細胞群3的“細胞組織”呈現了明顯的高表達。
布魯克timsTOF fleX質譜儀同時具備MALDI成像功能和LC-MS/MS組學分析功能,成功地實現了空間定位組學工作流程中的關鍵步驟。與傳統組學的“整體勻漿方法”相比,空間定位組學充分地考慮到乳腺癌組織中不同的腫瘤亞細胞群之間的差異性,并分別對各個腫瘤亞細胞群體進行了有針對性的、深度的蛋白組分析。以上的眾多優點使得空間定位組學為精準地揭示乳腺癌的致病機理奠定了堅實的基礎。