前沿顯微成像技術專題——超分辨顯微成像(2)
上一期我們為大家介紹了幾種主要的單分子定位超分辨顯微成像技術,還留下了一些問題,比如它的分辨率是由什么決定的?獲得的大量圖像數據如何進行重構?本期我們就來為大家解答這些問題。

單分子定位超分辨顯微成像的分辨率
單分子定位超分辨顯微成像的分辨率主要由兩個因素決定:定位精度和分子密度。定位精度是目標分子在橫向尺寸上的位置,分子密度是樣品中目標分子的密度。兩者在決定分辨率時同樣重要。
定位精度
在單分子定位超分辨顯微成像中,單像素通常被放大到100 nm左右,即光學衍射極限(200nm~250nm)的一半, 以滿足奈奎斯特雙倍采樣定律。超分辨成像的終極目標,是要對單分子實現達到10nm的亞像素定位,即在單個像素上能夠檢測到多個發光點。

圖1 通過高斯擬合的亞像素定位
亞像素定位一般是通過高斯擬合實現的。以光學衍射極限250nm為例,這個熒光點能覆蓋3x3的100nm像素陣列,如圖1。高斯擬合首先通過分析相鄰像素的強度,將最亮的像素定為中心像素,也就是發光點應該在的像素位置。如果中心像素左側像素的強度高于右側,高斯分布的峰值,也就是發光點必定位于中心像素左側。同理,如果中心像素上方的像素的強度高于下方,峰值必定位于上方。再結合從四個角的像素獲取的信息,就可以精確地確定發光點的峰值位置。
高斯曲線的高度和寬度與檢測到信號強度直接相關,信號越強,高斯曲線的峰值越高、形狀越細,定位就越準確。因此,提高定位精度最重要的就是增加檢測到的光子數。這就是單分子定位超分辨顯微成像需要選擇高效率的熒光基團和高靈敏度的相機的原因——高量子效率的背照式和大像元的相機能夠收集更多的光子。
不過要注意的是,如果相機的像素尺寸大于衍射極限的一半(100nm),高于衍射極限,像素尺寸就會成為定位精度的限制因素,反而會增加定位的不確定性。因此應當在確保滿足奈奎斯特采樣的前提下,盡量選擇大像元的相機。
另外,圖像背景會降低圖像對比度,也會降低定位精度。背景包括相機噪聲、散射光、非特定標記、自發熒光和樣品產生的其他噪聲源等等。要降低背景,除了在樣品制備過程中盡量減少背景熒光外,選用低噪聲相機也很重要。
分子密度
如果只是定位精度很高,但是卻沒有足夠多的目標分子來解析樣品結構,高定位精度也就失去了意義。根據奈奎斯特定理,兩個相鄰分子之間的距離應該是所需分辨率的兩倍。也就是說,如果要實現20nm的超高分辨率,每10nm就需要有一個目標分子,才能滿足采樣密度的要求。
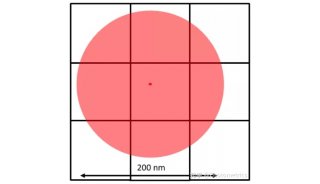
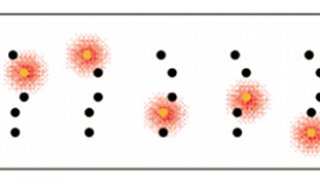
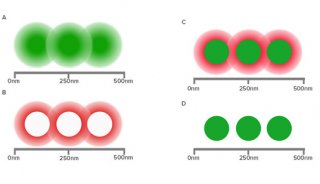
圖2 顯示了三個定位精度相同,但分子密度不同的模擬樣品的成像情況。最右邊的樣品有足夠的分子密度,因此能清楚的呈現出樣品邊緣位置,而最左邊的樣品完全不能解析樣品邊緣的特征。

圖2 超分辨率成像中的局部分子密度(From Zeiss Campus)
超分辨顯微定位重建算法
在獲得了信號強度和分子密度都足夠的高質量的原始數據后,還要靠重建算法,對檢測到的目標分子進行定位,并重建最終的超高分辨率圖像。
一般來說,重建算法需要完成以下7個步驟:
確定每個像素的噪聲水平
圖像平滑,去除背景熒光
找出每個目標分子的大致位置 (以上第1,2步預處理,就是服務于這個預定位)
對目標分子進行亞像素定位。這是實現超分辨的基礎,在每幀圖像上定位每一個目標分子的點擴散函數的峰值位置。
克服“密集場”問題,即多個目標分子存在于同一區域,需要單獨定位
后處理。通過一系列后處理對數據進行修正,包括去除定位不良的點,合并或刪除重復點,校正漂移等
重建完整的超分辨圖像
重建算法的設計并非一刀切,許多實驗室選擇根據不同的實驗自行設計不同的重建算法,以更好地匹配不同的熒光基團和樣本。即使是同一算法,也需要調整多個參數,才能獲得高質量的數據。
重建PALM和STORM的一個最常見的算法就是ThunderSTORM(ovesn_et al.,2014)。它是一個開源的交互式ImageJ插件,強大的功能可以滿足對超分辨定位數據進行復雜的分析,自由度高,方便研究人員使用。其他流行的PALM和STORM開源算法,如?RapidSTORM(Wolter et al.,2012) 和?QuickPALM(Henriques et al.,2010) 等,可以提供更高的數據重建速度。
對于DNA-PAINT來說,最流行的算法是Picasso,由Jungmann實驗室設計。它與其他重建算法具有相同的功能,也可以用于任何定位顯微技術。免費下載地址:https://www.biochem.mpg.de/5608779/Software
現在讓我們總結一下,進行單分子定位超分辨顯微成像,你需要:
高靈敏度,低噪聲的科學相機
高效率的熒光探針,適當(和定位精度相匹配)的標記密度
合適的重建算法
Teledyne Photometrics 的?Prime?系列背照式sCMOS相機(Prime 95B,Prime BSI)具有sCMOS相機的最高靈敏度,是單分子熒光和超分辨成像應用的理想選擇。它們還可配備PrimeEnhance?和PrimeLocate?智能 FPGA 圖像處理功能,可以提高超分辨成像的定位精度,幫助您輕松處理海量數據。
PrimeEnhance
我們都知道單分子熒光信號在理想狀態下是一個光點。然而,實際成像過程中噪聲這個如影隨形的幽靈會對定位精度產生很大的影響。通過適當的降噪算法,可以在樣品和成像條件不變的情況下,顯著提高圖像信噪比,達到提供定位精度的目的。
通過算法降低噪聲聽起來容易,事實上存在許多挑戰,如何保留數據的定量性,以及樣品的關鍵特征,如對比度較低的邊緣、紋理和細節等都是需要考慮的問題。除此以外,還必須保證降噪算法不會產生新的偽影,并能適用于不同類型的圖像。另一方面,由于散粒噪聲隨信號的變化而變化,許多降噪算法很難區分信號和噪聲,導致樣品中小的細節被去除。

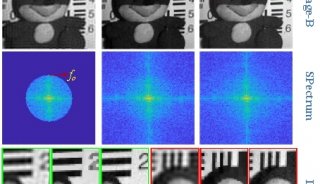
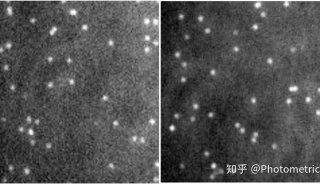
我們隆重向您介紹PrimeEnhance功能,它能夠在將圖像信噪比提高3-5倍的同時,保留樣本的細節和特征,適用于不同類型的圖像,并不引入偽影。更關鍵的是,它能確保處理后的圖像信號強度保持不變,從而不影響定量計算。從圖3a和3b的對比我們可以很明顯的看到,PrimeEnhance功能打開之后,信號強度不變,圖像信噪比顯著提高,還能呈現出之前分辨不了的樣品細節特征,簡直神器!您只需要在相機參數設置里打開PrimeEnhance功能,就可以獲得想要的高信噪比圖像啦!

圖3 開啟PrimeEnhance功能之前 (左) 和之后(右)圖像對比,下方為兩張圖中綠線對應的線強度曲線。
PrimeLocate
超分辨定位顯微成像要求單張圖像中,目標分子整體分布稀疏,以分辨出單個熒光分子。所以我們拍到的單張圖像大部分都是背景。如果能夠通過預處理將背景從圖像中刪除,獲取的數據量和數據處理所需的時間都會大大減少。
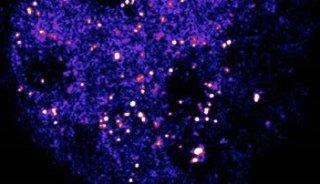
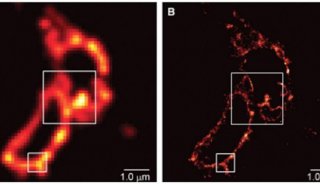
PrimeLocate?功能就是幫您去掉這些無用又占地方的背景的。它可以識別圖像中的單個分子,并僅將這些區域保存下來(圖4)。如果我們比較數據壓縮能力,不使用PrimeLocate,壓縮比約45%;使用 PrimeLocate,壓縮比可以增加到約85%。顯著減少所需的存儲空間,解放硬盤的同時還能讓您的 sCMOS 相機以更快的速度工作。

圖4 PrimeLocate能動態識別圖像中的單個分子(a),去除區域周圍的背景,僅傳輸單個分子的信息(b)
正所謂工欲善其事,必先利其器。想要得到高質量的圖片進行超分辨定位,相機的選擇是至關重要的!在下一期中,我們將對背照式 sCMOS 和 EMCCD 的成像效果進行直觀比較,看看誰才是這一領域的王者。敬請期待~
References
Henriques, R., Lelek, M., Fornasiero, E. F., Valtorta, F., Zimmer, C. & Mhlanga, M. M. (2010) QuickPALM: 3D real-time photoactivation nanoscopy image processing in ImageJ. Nat Methods. May;7(5):339-40. doi: 10.1038/nmeth0510-339.
Jungmann, R., Avenda?o, M. S., Woehrstein, J. B., Dai, M., Shih, W. M. & Yin, P. (2014) Multiplexed 3D cellular superresolution imaging with DNA-PAINT and Exchange-PAINT. Nat Methods. Mar; 11(3): 313–318. doi: 10.1038/nmeth.2835
Ovesny, M., K?í?ek, P., Borkovec, J., Svindrych, Z. & Hagen, G. M. (2014) ThunderSTORM: a comprehensive ImageJ plugin for PALM and STORM data analysis and super-resolution imaging. Bioinformatics. Aug 15;30(16):2389-90.
Wolter, S., L?schberger, A., Holm, T., Aufmkolk, S., Dabauvalle, M. C., van de Linde, S. & Sauer M. 2012. rapidSTORM: accurate, fast open-source software for localization microscopy. Nat Methods. Nov;9(11):1040-1.
-
科技前沿
-
項目成果