布魯克在美國HUPO 2021大會最新成果發布
——深度無偏向性血漿蛋白質組學、PaSER 1.1軟件和新型交聯耗材的新成果
牛津大學Roman Fischer教授展示了高通量4D-蛋白質組學技術,只需10~20分鐘即可在未去高峰度蛋白的血漿樣本中檢測出大于350種蛋白質,實現了高通量、高度可靠性的生物標記物發現。
OmicEra診斷集團實現了在單臺timsTOF Pro系統上,以每天60個樣品的分析速度,僅用時12天,實現了對源于31名COVID-19患者的720個未去高峰度蛋白的血清樣本檢測,成功定量分析了502種蛋白質,發現其中116種蛋白質在表達水平上具顯著變化。
使用Seer Proteograph?產品套件對血漿樣本處理后,在TIMS/PASEF分析時可得到前所未有覆蓋深度、高通量和高擴展性結果——鑒定出超過1,700種蛋白質。
PaSER軟件1.1版本正式發布,內容包括性能增強和4D-蛋白質組學在淌度輔助質量對齊(MOMA)分析的可視化。
推出了可富集的PhoX交聯劑和三個可裂解交聯劑,MaxQuant和XlinkX支持分析分析來自timsTOF的交聯數據,可用于結構蛋白質組學和蛋白質-蛋白質相互作用(PPI)相關研究。
2021年3月8日,布魯克在第17屆美國HUPO線上會議(US HUPO 2021)上宣布了深度、無偏向性血漿蛋白質組學方面等多項合作進展,這些合作展現了timsTOF Pro在4D-蛋白質組學研究中的高靈敏度和高動態范圍。
布魯克與科羅拉多大學安舒茲醫學中心Kirk Hansen教授的最新合作成果于美國東部時間3月9日下午在美國HUPO大會上進行的布魯克線上研討會中發表。Hansen教授在報告中介紹了COVID-19患者血漿蛋白質組學的研究,以及創傷患者血漿蛋白質組學變化的大隊列比較。
Hansen教授對每個樣品進行成千上萬次測量,揭示了急性損傷和疾病狀態下蛋白質的動態變化,通過開展更大范圍的臨床研究,團隊闡明了兩者之間的分子關系與機制,該研究具有超越當前診斷方法的潛力。
Hansen教授說:“因為timsTOF Pro的高掃描速度,高靈敏度和高通量的特性,我們最近才重新進入血漿蛋白質組學領域。結合Evosep One系統,兩者搭建的系統提供了幾年前不可能實現的高可靠性和高通量。”
2021年初,布魯克與Mann教授團隊發表的論文,突破性地展示了無偏差定量分析的真單細胞蛋白質組學,其可以解決單細胞生物學和病理生物學中的重要問題(https://www.biorxiv.org/content/10.1101/2020.12.22.423933v1)。布魯克計劃在2022年初推出單細胞專用的timsTOF系統,用于單細胞蛋白質組學的無偏差定量分析。
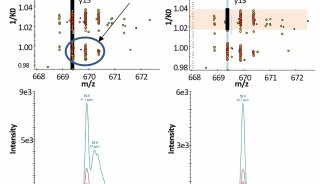
布魯克最近與Mann和Theis團隊共同在《Nature Communications》發表了最新成果,該研究涉及大規模肽段的碰撞截面(CCS)測量和4D-蛋白質組學機器學習的優勢(doi.org/10.1038/s41467-021-21352-8)。利用機器深度學習,可以預測來自任何生物體中,任何肽段的CCS值,充分利用額外的第四維CCS信息為更先進的4D-蛋白質組學工作流程奠定了基礎。
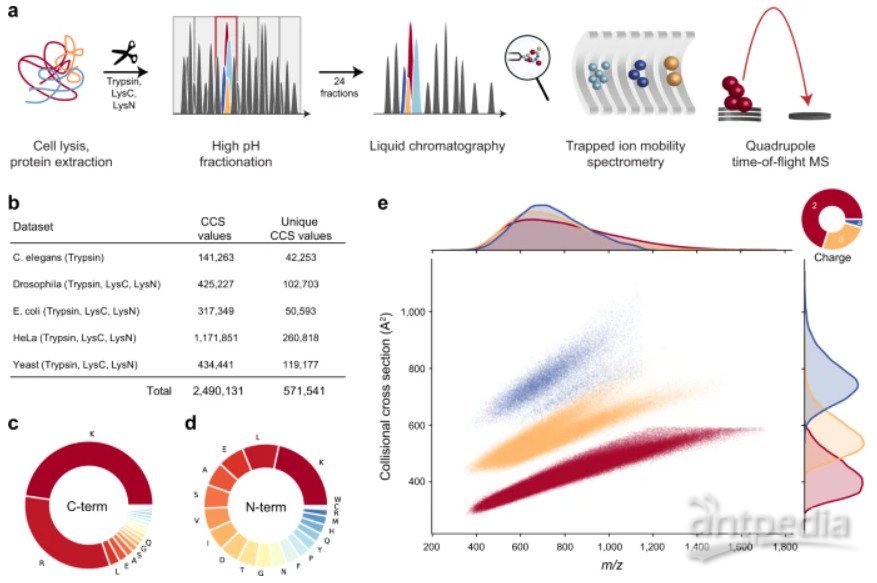
圖1:用TIMS和PASEF進行大規模肽段CCS測量
來源:www.nature.com/articles/s41467-021-21352-8
PaSER 1.1 實時蛋白質組學搜索
布魯克發布了PaSER軟件v.1.1,該軟件在3月9日美國HUPO研討會上,由布魯克公司蛋白質組學業務開發總監Chris Adams博士做了詳細介紹。PaSER是在布魯克收購的IP2軟件基礎上,基于GPU計算的蛋白質組學數據庫實時搜索軟件。PaSER是Parallel Search Engine In Real-time的縮寫,是在GPU上進行并行化、多線程的實時搜索,比常規的數據采集能更快地獲得結果。在實驗完成后,就可以“實時”獲得肽段和蛋白質的鑒別結果,實現高通量4D-蛋白質組學分析。

除了性能增強之外,PaSER 1.1還增加了4D數據的可視化,包括MOMA特征功能和搜索結果。4D-蛋白質組學分析的一個強大功能是淌度輔助質量對齊(MOMA)分析,在常規3D-蛋白質組學中無法區分的共洗脫同分異構體,利用精確的CCS值進行淌度分離。
化學交聯用于結構蛋白質組學和蛋白質-蛋白質間相互作用(PPI)研究
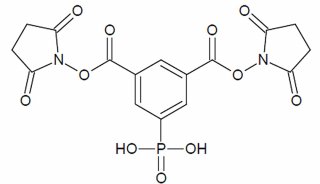
布魯克宣布推出用于研究蛋白質結構和相互作用研究的新款蛋白質化學交聯(XL-MS)耗材和軟件。在獲得烏特勒支大學許可后,由Albert Heck和Richard Scheltema團隊開發的PhoX交聯劑將于今年春季由布魯克進行商業化。PhoX是一種具有磷酸酯基團的可富集交聯劑,利用金屬磁珠,可以在XL-MS反應產生的復雜混合物中進行親和純化。這種富集極大地增強了檢測交聯肽的能力,Albert Heck、Richard Scheltema和布魯克之間的合作研究表明,通過使用caps-PASEF進行TIMS分離,可以顯著增強交聯肽段檢測能力(MCP,2020年7月20日,19(10):1677-1687)。布魯克在春季還將推出3種可裂解交聯劑,由于在MS/MS實驗中,交聯劑的裂解便于檢測到分子間的特征質量差異,該優勢使這3種交聯劑在某些研究小組中被列為首選。
化學交聯數據分析非常復雜,需要自動分析軟件。來自德國馬克斯·普朗克研究所的Juergen Cox教授團隊開發的新版MaxQuant軟件中,已經支持分析timsTOF Pro系統中XL-MS數據。目前團隊正在進行beta版測試,預計將于2021年4月發布。

深度、無偏向性血漿蛋白質組學的研究進展
最近,幾個研究小組使用多種方法對血漿蛋白質組學研究并得到了令人興奮的結果。2021年2月,牛津大學納菲爾德分校醫學院的Roman Fischer教授,在Genetic Engineer & Biotech News上發表了“High-Throughput Proteomics During a Pandemic”該項研究成果。Fischer教授使用高通量4D-蛋白質組學研究了數百個來自不同患病程度的COVID-19患者血漿樣品、健康人群以及患有其他疾病(如敗血癥)患者的血漿樣本。
同樣在2021年2月,來自德國Planegg的OmicEra診斷集團的研究人員在MedRxiv上發表了預印稿“High-resolution longitudinal serum proteome trajectories in COVID-19 reveal patients-specific seroconversion”。研究中31名樣本患者在31天里進行縱向追蹤,并使用高通量4D-蛋白質組學分析。這項研究采用OmicEra自動化蛋白質組學流程,搭配在Evosep One色譜系統上運行21分鐘的洗脫梯度,可達到每日60個樣本的分析通量。
使用這個方法,OmicEra在單臺timsTOF Pro系統上分析了720個未去高峰度的血清樣本,歷時12天,總共定量分析了502種蛋白質,發現約116種蛋白質在表達水平上發生了變化。在US HUPO上,布魯克將與OmicEra聯合發布血清蛋白質組學的相關應用說明。
Seer(www.seer.bio)在US HUPO 2021上展示了與布魯克合作成果的海報。海報展示了Seer制造的納米顆粒與TIMS/PASEF方法相結合的獨特分析功能。該研究使用90分鐘梯度的DDA nanoLC-TIMS-MS/MS方法鑒定出了1,700多個蛋白,實現了無偏差、高覆蓋深度、高掃描速度的血漿蛋白質組分析。Seer的Proteograph產品套件與TIMS/PASEF方法的結合,為無偏差、深層蛋白質組學提供了擴展性的解決方案,幾乎所有實驗室都可以使用。
布魯克蛋白質組學業務發展總監Chris Adams說:“我們很高興看到血漿蛋白質組學領域正在快速發展,我們期待該領域在液體活檢多組學轉化研究和生物標志物驗證中迅速發展。TIMS/PASEF方法的高通量、高穩定性以及4D-蛋白質組學獨特的選擇性和靈敏度等優勢,使血漿蛋白質組學在深層、無偏差分析方面取得了實質性的進步。”