如何設計PCR引物(一)
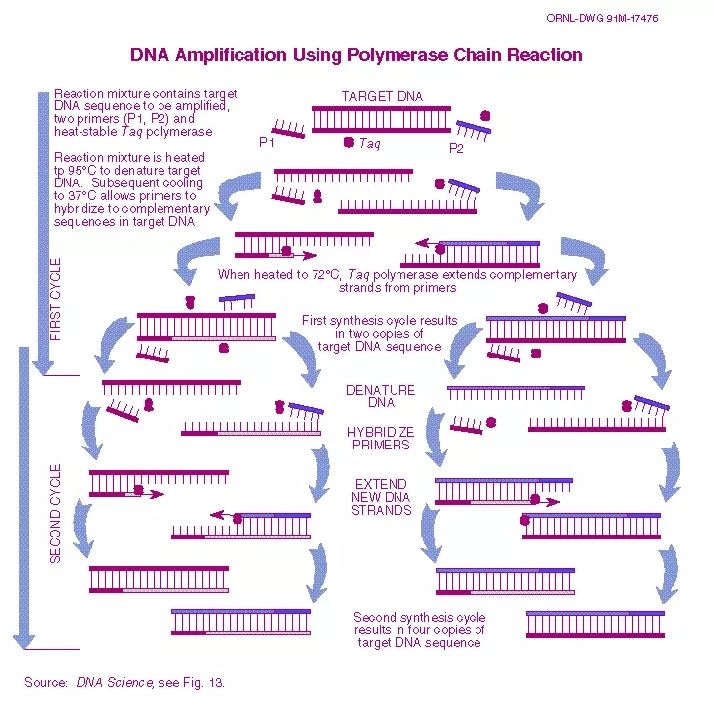
“如何設計PCR引物”看到這個題目后肯定有問“什么是PCR”的。對于這個問題,連小白都知道,您不知道,就只好去找百度哥咯。雖然百度哥肚子里其他的信息亂七八糟的,但是這個問題的答案還是蠻統一的,不會有誤導性,但問無妨。另外,也可以看看下圖老外給的簡介,英文不好請有道。
跟“服裝設計”類似,設計PCR引物也需要工具的。因此,在做設計之前,先翻翻自己的電腦是否有“primer premier”或者“Oligo“之類的引物設計專用軟件。當然,這些軟件都是破解版的。說到這,先喊下口號“支持正版,打擊盜版!”。如果您恰巧沒有,也不用急,請留下郵箱地址,我會悄悄發送給你。做PCR無非兩種目的:一是用于病原體檢測;二是用于生
物學研究,如克隆、測序等。根據目的不同,PCR引物設計的流程略有不同。
首先來講講如何設計PCR引物用于病原體檢測,這也是分子診斷常涉及的問題之一。對病原體的檢測,保證檢測特異性是關鍵。因此,在做設計之前,先得找特異性核酸序列。這些序列要相對保守,即突變率低。如小白要做的逆轉錄PCR——一類以RNA為目的序列的PCR,就得找RNA對應的cDNA(互補DNA,可問百度哥)保守序列來做引物設計。類似地,找保守序列也要用工具的,熊哥常用“Vector NTI Advance”里的“AlignX"模塊。當然,您也可以選擇相對簡單的DNAman軟件來做比對。如果您恰巧還是沒有這個軟件,沒關系,請留下郵箱地址吧。好了,下面開始第一個手把手演示——如何用AlignX來做序列比對。
手把手演示1:用AlignX做序列比對。本次演示以查找手足口病毒EV71的衣殼蛋白VP1基因保守序列為例。
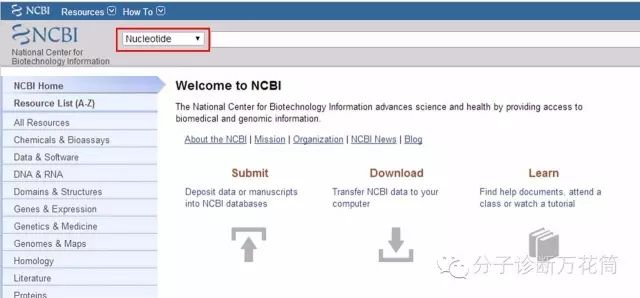
1. 登陸”http://www.ncbi.nlm.nih.gov“網站,選擇“Nucletide”。
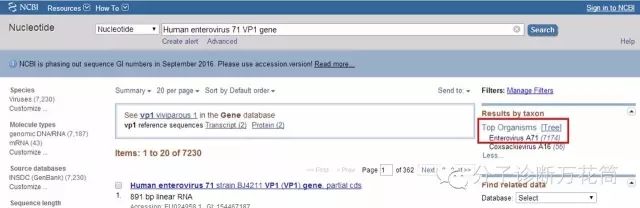
2. 在“Nucletide”空白欄里輸入關鍵詞,本實例輸入“Human enterovirus 71 VP1 gene”,然后在右側“Results by taxon”欄里選擇“Enterovirus A71”。
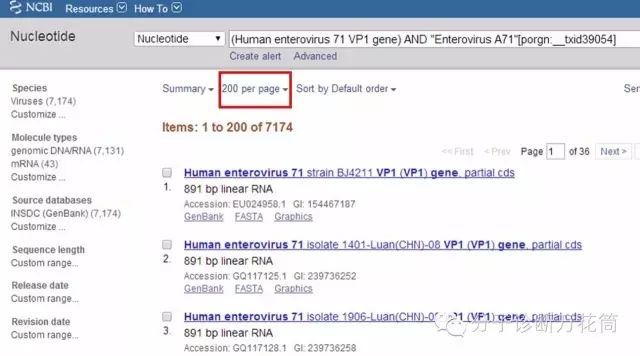
3.你會發現有7174條相關序列。一般系統默認每頁只顯示20條,當然可以設置顯示數為最大的每頁200條。
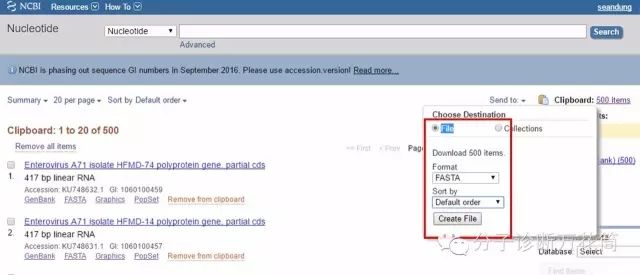
4.這時可以點擊“Send to”在Choose Destination界面中選擇“Clipboard”會將前500條序列加入Clipboard。
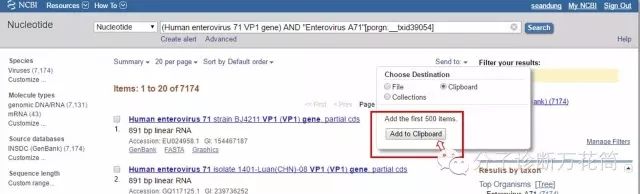
點擊右上角的“Clipboard: 500 items”,就意味著已經選擇了7174條中的500條,而這500條也是最新上傳的核酸序列。
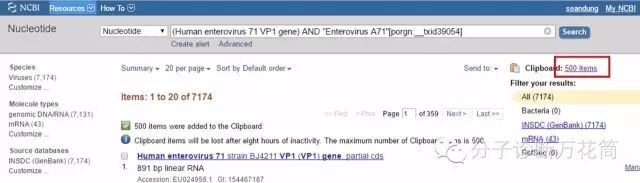
再點擊“Send to”在Choose Destination界面中選擇“File”,在format下選擇"FASTA",點擊“Create File”,會將前500條序列下載下來。
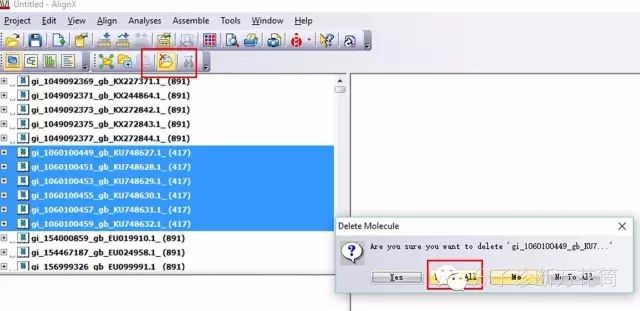
5.打開“AlignX”,點擊導入數據按鈕,選擇剛才下載下來的FASTA格式文件,即可導入序列。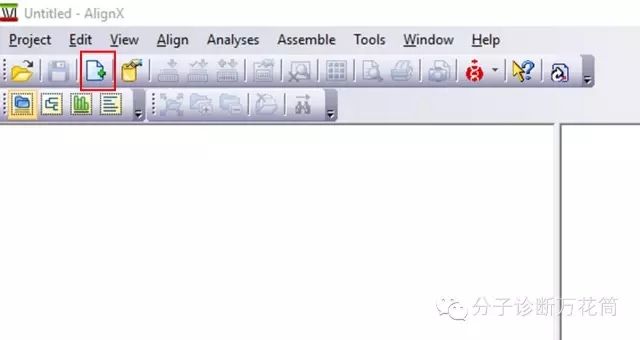
然后進行序列篩選,剔除那些過短的序列。本例中,完整的VP1基因有八百多bp,故低于八百的全剔除。
特別說明:如果想讓保守序列更加準確,比對序列的數量越多越好。當然,此時得考慮小白的電腦能不能承受,如果想Note7一樣發生自燃了,不要怪隆地熊哦。哈哈!
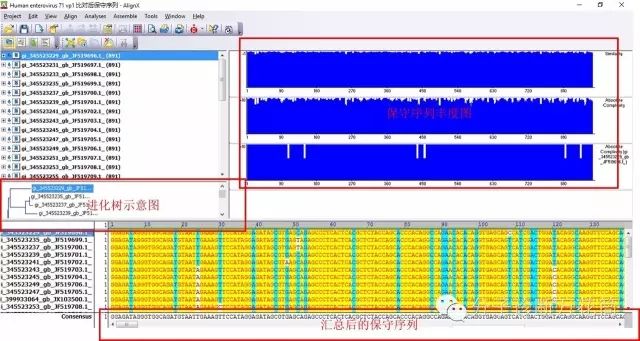
比對完后的結果是這樣的:
到此,小白要設計的PCR引物序列其3'大部分序列最好在這些黃色區域內,再也不用擔心漏檢了。當然,要保證特異性,還得進入第二個手把手演示了,如何利用保守序列設計PCR引物并驗證其特異性。