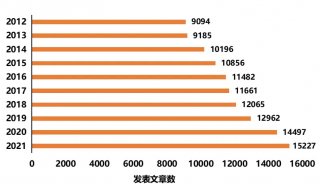
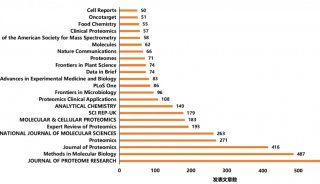
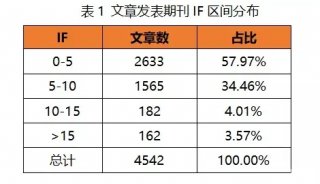
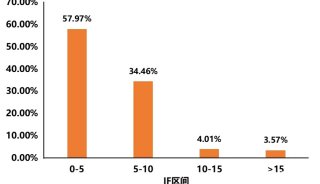
Nature Communications:尿液蛋白質組學研究新突破——標準操作流程(SOP)與全流程質控(QC)體系助力多平臺尿液蛋白質組學研究
2025年1月26日,北京航空航天大學醫學科學與工程學院劉超團隊和中國醫學科學院基礎醫學研究所孫偉團隊聯合北京理工大學、北京師范大學、中國科學院動物研究所、北京兒童醫院、北京協和醫院、國家蛋白質科學中心(北京)、中國科學院大連化學物理研究所、北京大學、昌平實驗室、深圳灣實驗室、廣州國家實驗室、Thermo公司、Bruker公司、SCIEX公司等共19個實驗室/課題組/公司的20個LC-MS平臺,在Nature Communications上發表了題為“Standard operating procedure combined with comprehensive quality control system for multiple LC-MS platforms urinary proteomics”的研究成果。


圖1 文章截圖
研究背景
尿液作為人體排泄物之一,含有豐富的生物信息,尤其是蛋白質信息。尿液蛋白質組學研究有助于發現敏感且非侵入性的生物標志物,對于疾病的早期篩查、診斷及預后評估具有重要意義。目前尿液蛋白質組學研究缺乏標準化體系,不同實驗室采用的尿液蛋白質組學分析流程和得到的鑒定深度差異較大,尿液生物標志物潛在價值尚未被充分挖掘,這些都限制了尿液蛋白質組學的廣泛應用。
研究方法
為了解決上述問題,研究人員開展了一項涉及20個LC-MS平臺的研究(圖2)。研究開發了MSCohort質量控制(QC)體系,提取81個指標用于單針實驗內和隊列實驗間數據質量評估和優化,并基于該QC體系建立了高通量尿液蛋白質組分析標準操作程序(SOP)。通過跨20個不同LC-MS平臺的尿液蛋白質組學數據采集和標準混合物種數據集驗證了該工作流程的穩健性、可重復性和定量準確性,最后將其應用于臨床大隊列結直腸癌尿液生物標志物的研究,發現了結直腸癌尿液生物標志物及其相關的生物學通路變化。
研究意義
這一研究成果為尿液蛋白質組學的大規模臨床研究提供了有力支持。通過統一SOP和全流程QC體系的應用,不同LC-MS平臺間的數據可重復性得到了顯著提升,為尿液蛋白質組學在精準醫學研究中的應用提供方法參考。

圖2 研究整體框架圖(來源: Nature Communications)
主要研究內容:
01 開發全面的蛋白質組學數據質量控制工具MSCohort
該研究全面總結和整合了現有的質控軟件提取的指標,并加入了26個新的質控指標,最終形成了81個質控指標的集合,可以實現對蛋白質組學數據的全面和綜合的指標提取和質控分析(圖3)。該研究開發的MSCohort質量控制工具可以支持DDA和DIA實驗的全流程評估和優化,以及DDA、DIA和PRM多個實驗間的穩定性分析和異常數據的篩選,同時生成全面的數據質量分析報告,幫助用戶快速評估實驗數據質量和定位實驗問題。

圖3 MSCohort軟件功能(來源: Nature Communications)
02優化和建立尿液蛋白質組學SOP
?????? 為了滿足大隊列臨床尿液蛋白質組學分析的需求,該研究優化和建立了高通量尿液蛋白質組學SOP。該SOP在樣本處理、色譜、質譜步驟都優化和整合了最優策略,包括高通量樣本處理方法、穩定高效的毛細管整體柱色譜體系、高靈敏度高通量的DIA采集方法,和全面系統的質量控制體系MSCohort進行實時數據質量評估(圖4)。采用此SOP可以實現:(1)單日可處理近200個尿液樣本,滿足大規模分析的需求;(2)在30分鐘的梯度內,單個尿液樣本中能鑒定出超過3000個蛋白質組(無需去除高豐度蛋白質);(3)實驗穩定性好,相同肽段在7天內的保留時間偏差小于0.2分鐘。該SOP在短梯度單次運行中提供了深度的尿液蛋白質組覆蓋度,為大規模隊列尿液生物標志物的研究奠定了良好基礎。

圖4 尿液蛋白質組學SOP的采用策略(來源: Nature Communications)
03 跨20個LC-MS平臺尿液蛋白質組學數據分析和對比
該研究在全國19個單位的20個LC-MS平臺進行了尿液蛋白質組采集和分析,涵蓋了Orbitrap、timsTOF和ZenoTOF等主流蛋白質組質譜平臺。其中10個實驗室采用統一的SOP(U01-U10),另外10個實驗室采用每個實驗室各自常用的樣本采集方法(M01-M10)。結果證明,當采用統一SOP后,不同LC-MS平臺產生的數據在尿液QC樣本中表現出更高的靈敏度和可重復性。不同平臺間鑒定數目一致性和蛋白質定量的Pearson相關性顯著提升(圖5)。

圖5 來自20個LC-MS平臺的尿液蛋白質組學數據分析結果(來源: Nature Communications)
04 多平臺蛋白質組定量和生物標志物檢測性能驗證
為了驗證統一SOP條件下不同平臺的蛋白質組定量和生物標志物檢測性能,研究團隊制備了混合物種標準數據集,其中Human、Yeast、E.coli在兩組中的理論比例分別為1:1,1:2,4:1。結果證明,在統一SOP的條件下,3個LC-MS平臺(Lumos,Exploris 480,timsTOF Pro 2)可以達到高度的鑒定一致性和定量準確性。其中Human蛋白質被認為是兩組間表達無差異蛋白質,Yeast和E.coli蛋白質被認為是兩組間表達有差異蛋白質。3個平臺的差異蛋白質判別性能AUC在0.93-0.99,具有較高的準確度(圖6)。

圖6 來自3個LC-MS平臺的混合物種數據集數據分析結果(來源: Nature Communications)
05應用于多平臺結直腸癌尿液蛋白質組研究
為了驗證以上尿液蛋白質組學SOP的泛化性能,研究團隊應用該SOP在不同平臺進行結直腸癌(Colorectal cancer,CRC)生物標志物研究。研究共包含80例CRC疾病樣本和80例健康對照(Health control,HC)樣本,分別在3個LC-MS平臺(Lumos,Exploris 480,timsTOF Pro 2)采集。結果顯示,在每個平臺鑒定尿液蛋白質總數均超過6600,在三個平臺鑒定尿液蛋白質總數目超過8000。三個平臺鑒定結果具有較高的一致性,差異蛋白質Pearson相關性大于0.95,并顯示出一致的疾病模式。最后,篩選了5個CRC生物標志物組合,在三個平臺的AUC值分別為0.933,0.881,0.894,均具有較高的判別性能(圖7)。

圖7 來自3個LC-MS平臺的結直腸癌隊列數據分析結果(來源: Nature Communications)
綜上所述,該研究開發了全面系統的蛋白質組學數據質量控制體系MSCohort和高通量尿液蛋白質組SOP,并驗證了不同LC-MS平臺產生的尿液蛋白質組數據表現出高靈敏度和高可重復性。為多中心、多平臺、高通量、大隊列樣本的尿液蛋白質組分析提供了質量保障。該研究為2024年倡議的中國尿計劃和世界尿計劃(World Urine Project)研究奠定了堅實基礎。
北京航空航天大學博士生劉響為論文第一作者,中國醫學科學院基礎醫學研究所孫海丹副研究員和北京航空航天大學博士生侯鑫行為共同第一作者。
該研究課題受到了科技部重點研發計劃、國家自然科學基金、中央高校基本科研業務費專項資金、北京市醫學研究機構公益類改革發展試點項目、中國醫學科學院醫學創新基金、北京市自然科學基金-大興創新聯合基金的資助和支持,項目得到了中國生物醫學信息中心,國家人口與健康科學數據共享平臺的支持和幫助。
?
文章全文:
Liu X, Sun H, Hou X, et al. Standard operating procedure combined with comprehensive quality control system for multiple LC-MS platforms urinary proteomics. Nature communications, 2025, 16(1): 1051. https://pubmed.ncbi.nlm.nih.gov/39865094/
-
焦點事件

-
精英視角

-
會議會展

-
綜述
-
焦點事件
-
科技前沿